Coronavirus, ecco la mappa 3D di una delle proteine di Sars CoV 2. Un aiuto per mettere a punto il vaccino
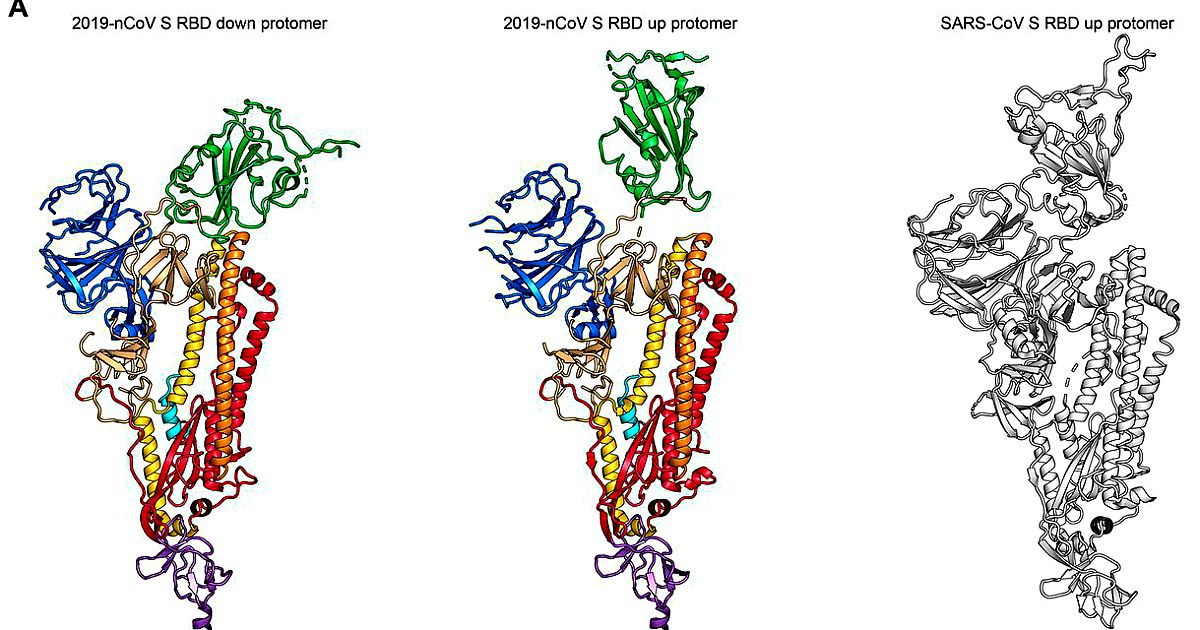
È la struttura molecolare di una delle armi che il virus usa per entrare nelle cellule. Pubblicato su Science, il risultato è stato ottenuto dall'ateneo con sede a Austin e dall’Istituto americano per la ricerca sulle malattie infettive (Niaid), parte dei National Institutes of Health (Nih)
Arriva dall’Università del Texas la mappa 3D del coronavirus Sars CoV 2 che potrà contribuire alla ricerca per una terapia e per un vaccino a cui sta partecipando la comunità scientifica internazionale. È la struttura molecolare di una delle armi che il virus usa per entrare nelle cellule. Conoscerla è importante per mettere a punto le armi per combatterlo. Pubblicato su Science, il risultato è stato ottenuto dall’ateneo con sede a Austin e dall’Istituto americano per la ricerca sulle malattie infettive (Niaid), parte dei National Institutes of Health (Nih).
La mappa riproduce nei dettagli la struttura di una delle proteine che si trovano sulla superficie del virus, chiamate ‘spike’ (punta o spina) e che funzionano come minuscoli grimaldelli che permettono al coronavirus di scardinare le porte d’ingresso delle cellule del sistema respiratorio umano per penetrare al loro interno e moltiplicarsi. Gli autori della scoperta, coordinati da Jason McLellan sono lo studente Daniel Wrapp e il ricercatore Nianshuang Wang, entrambi dell’Università del Texas. Tutti sono attualmente impegnati nella ricerca sul vaccino contro il nuovo coronavirus.
Fantastic that @sciencemagazine was able to get our manuscript online only days after @biorxivpreprint. Congratulations to Daniel, Nianshuang, @KizzyPhD and the rest of the team. We will continue to email coordinates to researchers, just email me. https://t.co/LKc1WTSrAr
— McLellan Lab (@McLellan_Lab) February 19, 2020
Conoscere la struttura della proteina significa avere un materiale prezioso per sollecitare la formazione di anticorpi. La molecola può inoltre essere utilizzata come una sorta di calamita molecolare per attrarre gli anticorpi presenti nel sangue di chi ha avuto l’infezione, raccoglierli e utilizzarli per combattere la malattia provocata dal virus, la Covid-19. Da anni, inoltre, il gruppo di MvLellan sta studiando altri coronavirus, compreso quello che nel 2015 ha scatenato la Mers, e il parente più stretto del Sars CoV 2, ossia il coronavirus che nel 2002-2003 è stato responsabile della Sars. Lo studio era stato pubblicato pochi giorni fa su BiorXiv e a stretto giro su Science.
Great effort led by grad student Daniel Wrapp and postdoc Nianshuang Wang! 12 days from harvesting media to deposition/submission. Now to test the stabilized spike as a vaccine with Dr. Barney Graham at VRC/NIAID. https://t.co/4Sj37RHL5t
— McLellan Lab (@McLellan_Lab) February 15, 2020
Per questo da tempo i ricercatori avevano messo a punto la tecnica che permette di rendere più facile e preciso lo studio della proteina, tanto da riprodurla appena due settimane dopo la pubblicazione della prima sequenza del virus da parte della Cina, all’inizio di gennaio; 12 giorni più tardi avevano ottenuto la struttura molecolare e avevano presentato l’articolo scientifico alla rivista Science per la pubblicazione; la rivista ha accelerato il processo di revisione da parte della comunità scientifica e ha anticipato la pubblicazione di un giorno, rispetto alla tradizionale cadenza settimanale di uscita. Èun esempio eloquente della corsa alla condivisione dei dati da parte di chi sta cercando di mettere a punto contromisure efficaci per combattere il nuovo virus.
L’articolo sul sito della Texas University